3 注解
启动子是DNA模板上专一地与RNA聚合酶结合并决定转录从何处起始的部位,也决定基因的转录效率。生物中有许多启动子,如大肠杆菌约有2000个启动子。各启动子的效率可不相同,大肠杆菌的强启动子每2秒钟启动一次转录,而弱启动子每10分钟才启动一次,从百多个大肠杆菌启动子结构的分析,得知两个强启动子的同源序列的中心在转录起始部位(基因编码链上第一个核苷酸) 5'侧约10和35个核苷酸处,弱启动子序列中往往有多处核苷酸被置换。许多原核生物都含有这两个重要的启动子区:
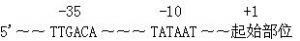
真核生物的启动子部位与原核生物不同,而且启动转录的活性,除需启动子外,还需某些外加序列。
启动子在遗传学中是指一段能使基因进行转录的去氧核糖核酸(DNA)序列。启动子可以被RNA聚合酶辨认,并开始转录。在核糖核酸(RNA)合成中,启动子可以和决定转录的开始的转录因子产成相互作用,继而控制细胞开始生产哪一种蛋白质。
完全的启动子称为规范序列。
4 启动子元件
启动子代表一些重要的元件可以与其他调节区域(如增强子、沉默子、边界元件或绝缘子)合作一致,以主导基因转录的水平。由于启动子一般都是在基因的上游,启动子所在的位置或是转录起始点会由+1开始编号。上游的位置所以都是由+1逆数的负数,例如-100就是位置100的上游碱基对。以下是各种启动子:
核心启动子是引发转录的必要部份及转录起始点,位置约为-35。且是RNA聚合酶的结合位点及一般转录因子结合位点。 近端启动子是基因的近端序列上游,包括一些基本的调控元件,位置约为-250,且是特定转录因子结合位点。 远处启动子是基因的远处序列上游,包括一些额外的调控元件,影响力较近端启动子弱。它是在上游更远的位置(但不是位置性的增强子或调控区域),是特定转录因子结合位点。
启动子规范序列的用途一般都是有问题的,且可引致对启动子序列的误解。在规范序列中,转录因子结合位点在特定细胞情况下有一个单独的序列会与蛋白质牢固地结合。但是自然选择会偏向较低能量的结合,作为一种调节转录输出。这种最普遍的序列称为野生型序列。这纵然不是最有利的序列,最近证据显示多种基因(包括原癌基因)都有G四股结构作为潜在调控信号。
在演化生物学的一个主要问题是修补启动子序列在演化过程中的重要性,例如人类血统从黑猩猩分开后的改变。某些演化生物学家建议启动子的演化或调节区域可能比序列编码更重要。
5 启动子序列
5.1 原核生物启动子
在原核生物中,启动子包含两个短序列位于从转录起结点起计的-10及-35上游位置。位于-10的序列称为普里布诺框或-10元件,及通常包含6个核苷TATAAT。普里布诺框在开始转录是绝对必要的。其他位于-35的序列通常包含6个核苷TTGACA。它的出现可以帮助非常高的转录率。一些启动子含有所谓的“延伸-10元件”(同源序列5'-TGNTATAAT-3'),从这些元件开始,-35元件在转录时显得不重要。上述的启动子结构只有以原核生物RNA聚合酶的σ-70形式来辨认。原核生物RNA聚合酶复合物连同其他σ因子可以辨认不同的核心启动子序列。
| 序列 | 核苷 | |||||
|---|---|---|---|---|---|---|
| -10序列 | T | A | T | A | A | T |
| 77% | 76% | 60% | 61% | 56% | 82% | |
| -35序列 | T | T | G | A | C | A |
| 69% | 79% | 61% | 56% | 54% | 54% | |
5.2 真核生物启动子
真核生物启动子是极端的分化及很难表现其特征。它们一般处于基因的上游及有着远离转录起始点的调控元件。转录复合物可以引起去氧核糖核酸(DNA)向自己屈曲,以容许放置调控序列。很多真核生物启动子,但不是全部,都包含一个TATA盒(序列TATAAA)会与TATA结合蛋白结合,以协助形成RNA聚合酶转录复合物。[1]TATA盒一般会处于非常接近转录起始点(通常于50个碱基对以内)。
真核生物启动子调控序列一般与转录因子结合,当中涉及形成转录复合物。一个例子是E盒(序列CACGTG),它会与碱性-螺旋-环-螺旋(bHLH)的转录因子结合。
7 与启动子功能变异有关的疾病
以下是从人类孟德尔遗传学(OMIM)证实与启动子故障有关,不论是因启动子序列直接突变或是转录因子或转录共激发因子的突变。而多种癌症都没有列下是因为从染色体易位产生嵌合基因:
要留意的是在病原学上大部份的疾病都是异质的,而在分子层面上一种疾病往往是指多种疾病,纵然它们的病征及治疗方法一致。疾病对治疗有不同的反应,是因背后分子源头的差异,这会是药物遗传学的范畴。
8 参考
Smale ST, Kadonaga JT (2003). "The RNA polymerase II core promoter". Annu Rev Biochem 72: 449-479. PMID 12651739.
Levine M, Tjian R (2003). "Transcription regulation and animal diversity". Nature 424 (6945): 147-151. PMID 12853946.
Hobbs, K.; Negri, J.; Klinnert, M.; Rosenwasser, L.J.; and Borish, L. (1998). "Interleukin-10 and transforming growth factor-beta promoter polymorphisms in allergies and asthma". Am J Respir Crit Care Med 158 (6): 1958-1962. PMID 9847292.
Burchard, E.G.; Silverman, E.K.; Rosenwasser, L.J.; Borish, L.; Yandava, C.; Pillari, A.; Weiss, S.T.; Hasday, J.; Lilly, C.M.; Ford, J.G.; and Drazen, J.M. (1999). "Association between a sequence variant in the IL-4 gene promoter and FEV(1) in asthma". Am J Respir Crit Care Med 160 (3): 919-922 id = PMID 10471619.
Kulozik, A.E.; Bellan-Koch, A.; Bail, S.; Kohne, E.; and Kleihauer, E. (1991). "Thalassemia intermedia: moderate reduction of beta globin gene transcriptional activity by a novel mutation of the proximal CACCC promoter element". Blood 77 (9): 2054-2058 id = PMID 2018842.
Petrij F, Giles RH, Dauwerse HG, Saris JJ, Hennekam RC, Masuno M, Tommerup N, van Ommen GJ, Goodman RH, Peters DJ, et al. (1995). "Rubinstein-Taybi syndrome caused by mutations in the transcriptional co-activator CBP". Nature 376 (6538): 348-351. PMID 7630403.

